Российские генетики и антропологи собрали коллекцию генотипов и фенотипов, обследовав 300 человек из разных регионов Северной Евразии. На этой базе данных они протестировали европейскую систему для предикции цвета глаз и волос по ДНК — HIrisPlex-S, и выяснили, что в популяциях Северной Евразии точность ее работы несколько ниже, чем в Западной Европе.
Проведя экзомное секвенирование, дополнительно включившее интроны 53 генов, вовлеченных в контроль пигментации, ученые нашли семь SNP, которые с высокой точностью предсказывали цвет глаз в изученной выборке, из них пять SNPне были описаны ранее. Для цвета волос они нашли 11 информативных SNP, из них восемь новых. По-видимому, в популяциях Северной Евразии с пигментацией глаз и волос связан несколько иной спектр аллелей, чем в населении Западной Европы. Очевидно, точность предсказания фенотипа по ДНК для популяций Северной Евразии повышается, когда к стандартной панели добавляются эти новые маркеры.
Определение вероятного цвета глаз и волос индивида по его ДНК — важная часть работы генетиков-криминалистов. Этой возможностью пользуются и палеогенетики, исследующие древнюю ДНК. Известны несколько генов, полиморфные участки в которых влияют на пигментацию волос и глаз и на пигментацию кожи, они были найдены в исследованиях геномных ассоциаций (GWAS). Наиболее важные из них включены в панель HIrisPlex-S. В эту панель входят 24 маркера (SNP и инделы), позволяющие предсказывать по ДНК цвет волос и глаз, и 17 маркеров для предсказания цвет кожи. Панель HIrisPlex-S хорошо работает для индивидов европейского происхождения, поскольку система HIrisPlex-S была разработана на европейских популяциях (главным образом, на голландцах). Проверка же ее на популяциях других регионов мира практически не проводилась.
Конечно, для большинства популяций за пределами Европы характерны только коричневые глаза и темные волосы, поэтому для них задача предсказания пигментации не стоит. Но существует и меньшинство – в некоторых популяциях Северной Евразии (например, некоторые группы на Алтае, на Кавказе) встречается фенотип со светлыми глазами и светлыми волосами. Генетически эти популяции не состоят в близком родстве с населением Западной Европы. Поэтому вполне возможно, что они имеют аллели пигментации, которые не входят в систему HIrisPlex-S, но обеспечивают у них фенотип светлых глаз и волос. Если это так, то эти неизвестные еще аллели имеют важное значение для криминалистических исследований в России.
Этой проблеме посвящена статья коллектива генетиков и антропологов из Института общей генетики РАН, Медико-генетического научного центра, МГУ им. М.В.Ломоносова и Института этнологии и антропологии РАН при участии коллег из других научных организаций в Москве, Краснодаре и Нурсултане (Казахстан), ведущий автор статьи – д.б.н., проф. РАН Олег Балановский, зав. лабораторией ИОГен РАН и г.н.с. МГНЦ. Статья опубликована в журнале BMC Genomics. Эта работа была проведена в рамках выполнения проекта Союзного государства «ДНК-идентификация». Была поставлена задача оценить эффективность системы HirisPlex-S в популяциях Северной Евразии, а также найти новые аллели, связанные с пигментацией в этих популяциях и оценить их влияние на цвет глаз и волос.
Для решения этой задачи была использована коллекция Биобанка Северной Евразии: взяты образцы ДНК от 300 человек, для которых также имелись качественные антропологические фотографии. Эти 300 человек были отобраны из 48 популяций, принадлежащих к коренным народам России и сопредельных стран. Популяции относились к четырем регионам: Европейская Россия, Западная Сибирь, Кавказ и Северная Азия (этот регион в данной работе включал и Центральную Азию).
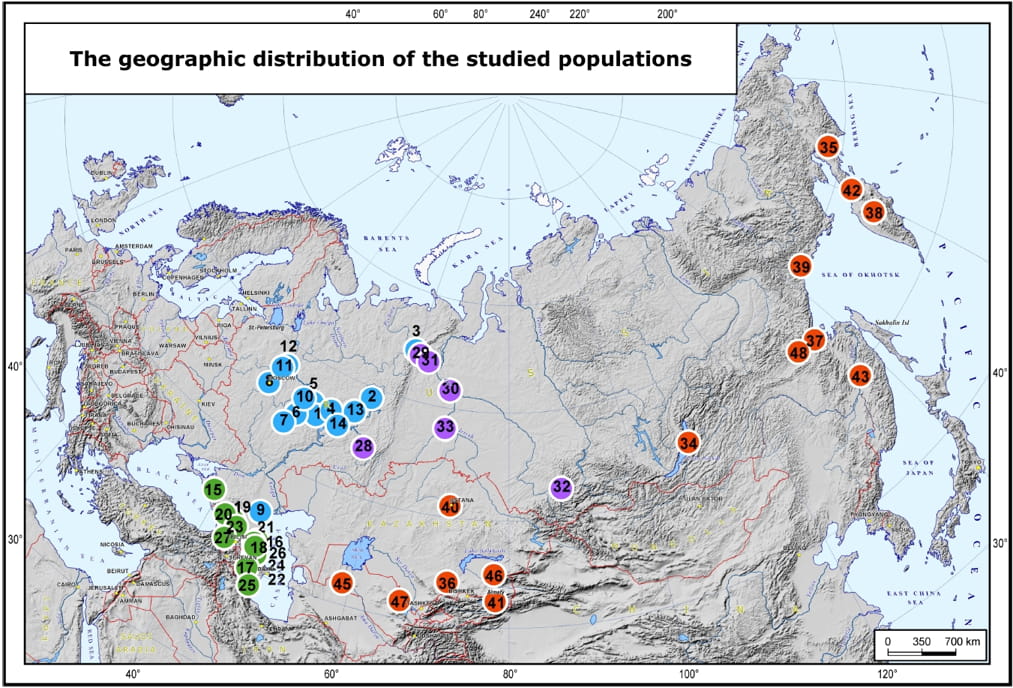
Расположение исследованных популяций на карте. 1 – чуваши, 2 – коми-пермяки, 3 – коми-зыряне, 4 – марийцы равнинные, 5 – марийцы горные, 6 – мордва эрзя, 7 – мордва мокша, 8 – русские, 9 – русские (казаки-некрасовцы), 10 – русские из Нижегородской области, 11 – русские из Тверской области, 12 – русские из Ярославской области, 13 – удмурты, 14 – татары Поволжья, 15 – адыгейцы, 16 – аварцы, 17 – азербайджанцы, 18 – даргинцы, 19 – кабардинцы, 20 – карачаевцы, 21 – кумыки, 22 – лезгины, — 23 – осетины, 24 – рутульцы, 25 – талыши, 26 – цахуры, 27 — турки месхетинцы, 28 – башкиры, 29 – лесные ненцы, 30 – ханты, 31 – манси, 32 – шорцы, 33 – сибирские татары, 34 – буряты, 35 – чукчи, 36 – дунгане, 37 – эвенки Дальнего Востока, 38 — эвенки Камчатки, 39 – эвенки Охотского побережья, 40 – казахи, 41 – киргизы, 42 – коряки, 43 – нанайцы, 44 – таджики, 45 – туркмены, 46 – уйгуры, 47 – узбеки, 48 – якуты Дальнего Востока.
Образцы ДНК изучили путем экзомного секвенирования. В отличие от широко используемого широкогеномного генотипирования, секвенирование позволяет анализировать не только известные аллели, но и обнаружить новые аллели, которые ранее не были найдены при GWAS. Поскольку многие уже известные ключевые SNP были расположены в интронах, авторы разработали специальную панель, которая кроме полного экзома включает также интронные и межгенные участки тех 53 генов, для которых ранее была показана их вовлеченность в генетический контроль пигментации. Определение цвета глаз и волос по фотографиям проводилось независимо тремя экспертами. Была создана объединенная база данных генотипов и фенотипов, а также региональные базы по четырем регионам.
На графике главных компонент авторы показали генетическое разнообразие изученных популяций вместе с популяциями, на которых была протестирована панель HirisPlex-S (голландцы, поляки, ирландцы и греки). Как видно на графике, генофонды популяций HirisPlex-S гомогенны и почти не перекрываются с популяциями, изученными в этой работе.
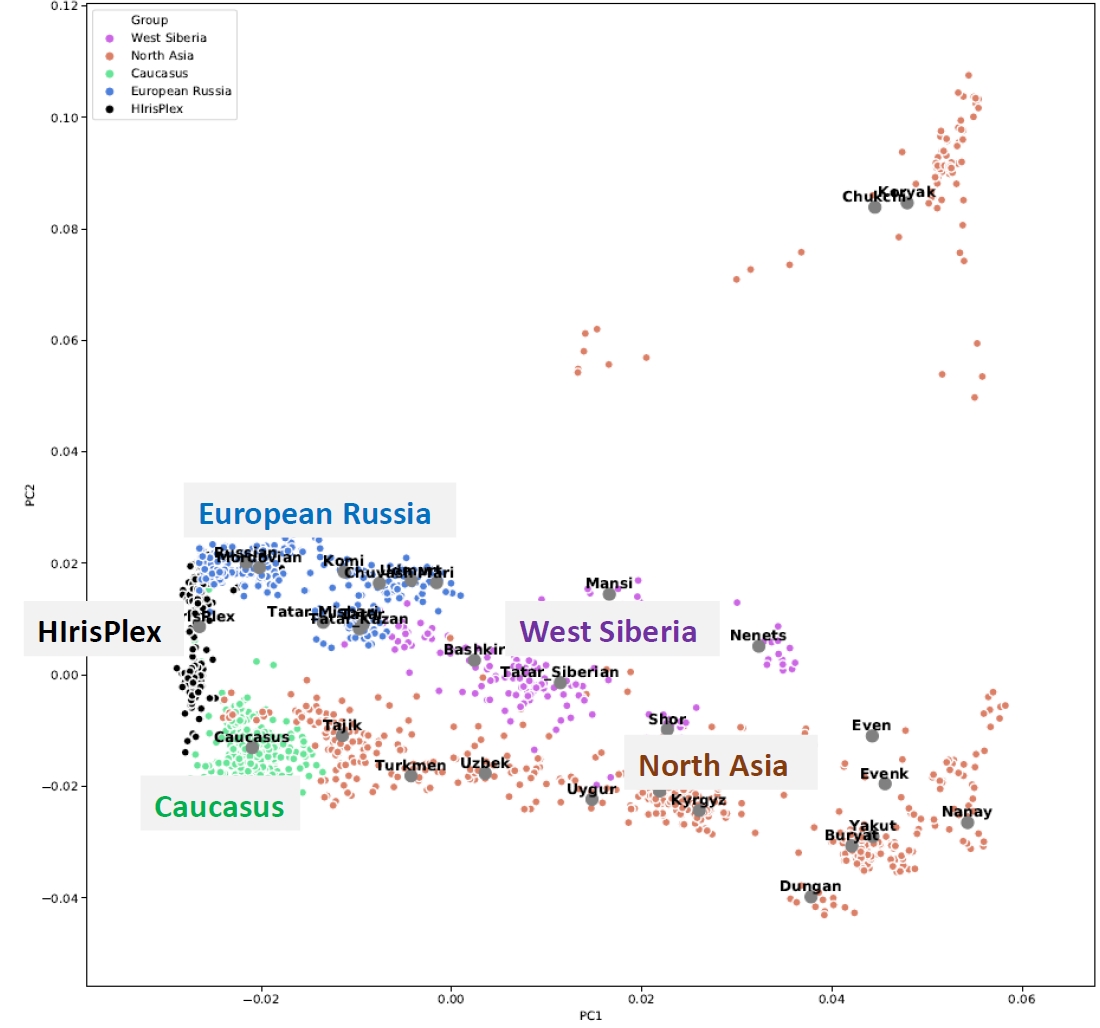
График анализа главных компонент. Региональные базы геномных данных обозначены цветными точками, цвет точек соответствует цвету региона: Европейская Россия – синий, Кавказ – зеленый, Западная Сибирь – фиолетовый, Северная Азия – темно-красный. Популяции Западной Европы, тестированные по системе HIrisPlex-S, обозначены черными точками.
Тестирование системы HirisPlex на популяциях Северной Евразии
Используя объединенную базу данных по всем регионам Северной Евразии, извлекли генотипы 24 SNP, включенных в HIrisPlex-S, сделали предсказание по этой панели, и сравнили предсказанные фенотипы с реальными. Таким способом оценили эффективность, с которой панель HIrisPlex-S предсказывает эти фенотипы как в объединенной базе данных, так и в разных регионах (за исключением Северной Азии, где частота светлых глаз очень низка). Точность предикции оценивали по величине AUC (площадь под ROC кривой), чем выше AUC, тем лучше работает классификатор. Оказалось, что эффективность HIrisPlex-S по предсказанию цвета волос и глаз в популяциях Северной Евразии несколько ниже, чем в популяциях Западной и Центральной Европы. Когда результаты проанализировали по каждому региону отдельно, оказалось, что предикция цвета глаз особенно снижена на Кавказе. Это может означать, что гены метаболических путей, вовлеченных в пигментацию глаз, в популяциях Кавказа несут иной спектр аллелей, чем в популяциях Западной Европы.
Поиск новых информативных аллелей в популяциях Северной Евразии
Используя данные расширенного экзомного секвенирования по исследуемым индивидам, авторы обнаружили 117 012 SNP в изучаемых ими 53 генах и межгенных регионах. Для всех этих SNP была проанализирована их связь с разными фенотипами пигментации. Этот анализ был проведен по пяти базам данных: объединенной базе по Северной Евразии и четырем региональным базам – по Европейской России, Кавказу, Западной Сибири и Северной Азии (только для анализа цвета глаз исключили базу по Северной Азии, так как по цвету глаз в населении этого региона практически нет изменчивости).
Для поиска новых информативных SNP проводили процедуру отбора признаков (feature selection) с использованием трех алгоритмов: f-регрессия, множественная инфо-регрессия, отбор признаков Lasso с разной степенью альфа (0.7, 0.5, 0.2, 0.1, 0.05, 0.01, 0.005, 0.001, 0.0005). На основании результатов этих трех алгоритмов отбора признаков, из всех SNP были отобраны топовые SNP для каждой базы данных.
В список SNP, информативных для цвета глаз, по объединенной базе Северной Евразии вошли 256 SNP. Для сужения этого списка каждому SNP был присвоен балл от 0 до 3 в зависимости от показателей перечисленных выше алгоритмов. В итоговый список лучших SNP вошли 36 SNP, получившие ненулевые баллы, они и использовались для создания классификатора. Из них высший балл 3 получили пять SNP. Два из них хорошо известны как связанные с цветом глаз (rs1129038 и rs12913832), остальные три не были описаны ранее.
Такой же анализ провели и по региональным базам трех регионов: Европейская Россия, Кавказ и Западная Сибирь. При суммировании анализов по объединенной и региональным базам 7 SNP получили наивысшие баллы и вошли в более чем одну базу. Два из них (rs1129038 и rs12913832) уже содержались в панели HIrisPlex-S, а остальные пять – кандидаты на новые сайты предикции цвета глаз в популяциях Северной Евразии. Авторы оценили частоту встречаемости этих пяти SNP в популяциях и выяснили, что они относятся, скорее, к обычным, чем к редким вариантам.
Итак, по итоговым результатам, минимальный набор для предикции цвета глаз в популяциях Северной Евразии содержит 7 SNP, два из которых входят в панель HIrisPlex-S, а значимость остальных пяти показана впервые. Оптимальный набор включает 36 SNP. По этим SNP авторы разработали классификаторы для предикции цвета глаз, используя алгоритм линейной регрессии. На изученных выборках классификатор на основе 7 SNP показал такую же точность, что и панель HIrisPlex-S из 41 SNP, а точность классификатора из 36 SNP оказалась несколько выше, чем точность HIrisPlex-S из 41 SNP. Из семи важнейших SNP шесть оказались локализованы в гене HERC2, а один (rs4812447) – в межгенном регионе. Ген HERC2 принадлежит к семейству генов HERC, которые кодируют группу больших белков, содержащих множественные структурные домены. Генетические вариации в этом гене ассоциированы с вариабельностью пигментации кожи/волос/глаз.
Такой же анализ провели для отбора SNP, информативных для цвета волос в объединенной базе Северной Евразии. Список из 322 SNP затем сузили до 33 лучших SNP, получивших ненулевые баллы. В список маркеров с наибольшей предиктивной силой вошли 11 SNP, получившие максимальный балл 3; три из них включены в панель HIrisPlex-S (rs16891982, rs12913832, и rs1129038), для остальных восьми связь с пигментацией ранее не была описана. Эти 33 SNP и 11 SNP послужили для создания классификатора по цвету волос, который различает четыре независимых цвета: рыжие, светлые, темные и смешанные.
Итак, авторы исследования проанализировали корреляции генотипов и фенотипов в популяциях Северной Евразии, которые имеют фенотипы светлой пигментации, но генетически отличаются от популяций Западной Европы. Выяснилось, что эффективность системы HIrisPlex-S для этих популяций несколько ниже, чем для западноевропейцев, для которых этот классификатор был разработан. Такое снижение точности может быть результатом популяционно-специфичных SNP, которые имеются в Северной Евразии, но не вошли в панель HIrisPlex-S. В работе были обнаружены пять новых маркеров, которые по точности предикции цвета глаз в популяциях Северной Евразии сравнимы с двумя важнейшими ранее известными SNP. Четыре из пяти новых маркеров лежат в гене HERC2, а пятый расположен в межгенном регионе. Эти SNP имеют достаточно высокую частоту в большинстве изученных популяций. Очевидно, точность предсказания цвета глаз и волос по ДНК индивида в популяциях Северной Евразии будет выше, если добавить в используемую панель эти маркеры. В будущих исследованиях предлагается протестировать эти новые SNP на дополнительных выборках из Северной Евразии.
Источник
Balanovska E., Lukianova E., Kagazezheva J., Maurer A., Leybova N., Agdzhoyan A., Gorin I., Petrushenko V., Zhabagin M., Pylev V., Kostryukova E., Balanovsky O. Optimizing the genetic prediction of the eye and hair color for North Eurasian populations // Balanovska E., Lukianova E., Kagazezheva J., Maurer A., Leybova N., Agdzhoyan A., Gorin I., Petrushenko V., Zhabagin M., Pylev V., Kostryukova E., Balanovsky O. Optimizing the genetic prediction of the eye and hair color for North Eurasian populations // BMC Genomics 2020 21(Suppl 7):527 https://doi.org/10.1186/s12864-020-06923-1